Génomique comparative et biochimie des mécanismes fongiques impliqués dans la symbiose mycorhizienne et les processus de décomposition du bois
Responsables scientifique : Francis Martin & Eric Gelhaye (UMR 1136 Interactions Arbres/Micro-organismes)
Partenaires Labex : Philippe Gérardin (Laboratoire d’Etudes et de Recherche sur le Matériau Bois LERMAB)
Collaborations : Professeur David Hibbet (Clark University), Dr. Igor Grigoriev (Fungal Genomics Program, U.S. DOE Joint Genome Insitute, JGI)
________________________________________________
Contexte — La lignocellulose est la principale source de biopolymères des écosystèmes terrestres. Sa dégradation (et donc le recyclage de ce puits de carbone) est particulièrement étudiée du fait de ses nombreuses applications potentielles comme la production de biomolécules, bioéthanol ou enzymes par exemple. Cette dégradation est principalement réalisée par des champignons et notamment des agaromycetes saprophytes selon deux mécanismes distincts permettant de différencier les champignons dits de pourriture brune de ceux dits de pourriture blanche. Les champignons ectomycorhiziens, qui jouent un rôle écologique important en tant que symbiotes des arbres, appartiennent également aux agaricomycetes.
Objectifs — Nous proposons d’étudier une cinquantaine de génomes d’agaricomycetes, saprophytes et ectomycorhiziens, représentatifs des évolutions indépendantes des traits de vie observées au sein de cette classe, dans le but d’identifier les réseaux de gènes impliqués dans l’adaptation de ces champignons à leurs niches écologiques. Nous étudierons également l’impact du polymorphisme génomique intra-espèce sur l’adaptation à son environnement de l’agaricomycete lignivore : Trametes versicolor
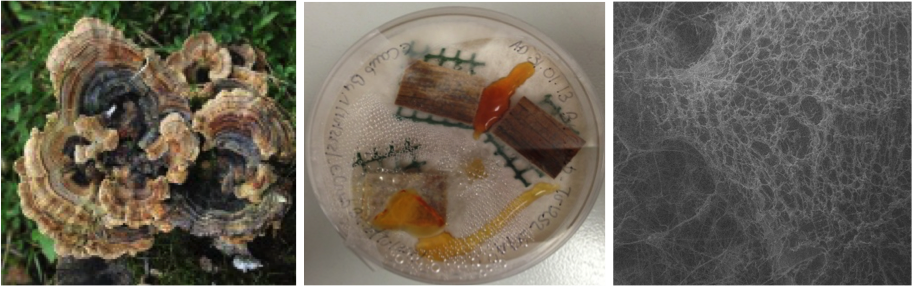
Démarche — Par génomique comparative, nous avons caractérisé différentes familles de gènes impliqués dans la dégradation de la lignocellulose chez les champignons lignivores et ectomycorhiziens (i.e., enzymes de dégradation des polysaccharides de la paroi végétale, oxidoreductases impliquées dans la dépolymérisation de la lignine). Afin de caractériser étudier le polymorphisme génétique intraspécifique, nous avons comparé les séquences génomiques de 10 souches de Trametes versicolor prélevées sur différents types de bois en décomposition.
Résultats marquants —
- L’analyse phylogénomique de 28 génomes de champignons ascomycètes et basidiomycètes a permis de mettre en évidence une forte corrélation entre le répertoire de gènes impliqués dans la décomposition de la lignocellulose et les traits de vie (pourriture blanche, pourriture brune et symbiote mycorhizien).
- Dix souches de Trametes versicolor isolées sur différentes essences d’arbres et de différentes origines géographiques ont été étudiées d’un point de vue phénotypique (vitesse de colonisation sur différents milieux, capacité à dégrader différentes essences de bois (chêne, hêtre, épicea), détection de différentes activités enzymatiques impliquées dans la dégradation de la lignocellulose sécretées durant la colonisation d’aubier de chêne, analyse de ces mêmes sécrétomes par protéomique). Une analyse en composante principale de ces données suggère la présence de deux groupes au sein de cette collection, groupes reliés à l’essence d’origine.
- L’analyse du polymorphisme des séquences génomiques (SNP) de cinq souches de T. versicolor semble confirmer la présence de ces deux écotypes corroborant ainsi les analyses phénotypiques.
Perspectives —
- Compléter l’analyse phylogénomique afin de dater les stades de l’évolution des différents traits de vie ;
- Poursuivre la caractérisation phénotypique et génomique des différentes souches de T. versicolor ;
- Caractérisation fonctionnelle des gènes/protéines impliqués dans l’adaptation des souches de de T. versicolor à leur environnement – le bois en décomposition ;
- Définir des marqueurs pour des approches de génétique des populations à corréler avec des données environnementales de manière à établir les relations potentielles entre génotypes et environnement (notamment structure chimique du bois).
Valorisation —
- Aurélie Deroy : Ecole doctorale RP2E (Poster)
- IRG International Research Group on Wood Protection (St George, Utah, USA, mai 2014)
- International Conference “the Plant and its Environment”, IFR 87, octobre 2013
- European Conference on Fungal Genetics (Seville, mars 2014)
- Whetzel-Westcott-Dimock Endowed Lectureship 2014, Cornell University, avril 2014
- 33th New Phytologist Symposium « Networks of Power & Influence », Zürich, mai 2014.
- XVI Molecular Plant-Microbe Interactions Congress, Rhodes, juillet 2014
- International Mycological Congress, Bangkok, août 2014
Projets soumis —
- Project JGI CSP WoodLeafLitterOME : coordination F. Martin, février 2014
- Project Screenzymes : coordination E. Gelhaye, février 2014