Développement de modules protéiques (PROTeic CARriers) pour la modification de fibres végétales
Responsable scientifique : Eric Gelhaye (UMR 1136 « Interactions Arbres/Micro-organismes » – IAM)
Partenaires LabEx : Philippe Gérardin (LERMAB)
Collaboration : C. Segovia (CETELOR), N. Attenot (BIOLIE)
_________________________________________________________
Contexte — La connaissance fine de l’organisation structurale et chimique de la paroi végétale, outre un enjeu de recherche fondamentale, constitue un point clé pour le développement de différentes biotechnologies liées à la chimie du végétal : bioraffinerie, industrie textile, biotechnologie blanche, biocomposites, extraction de molécules d’intérêt,… Cette variabilité est à corréler avec celle des systèmes enzymatiques mis en jeu par les micro-organismes pour dégrader puis minéraliser les différents constituants de la paroi végétale. Les micro-organismes (champignons et bactéries) sécrètent en effet une multitude d’enzymes permettant le fractionnement de la paroi et présentant souvent une spécificité vis à vis des différents constituants. De nombreuses enzymes impliquées dans la dégradation des carbohydrates (cellulose, hémicelluloses, pectines,…) sont modulaires et présentent un module de fixation à leur substrat (Carbohydrate binding module) et un module catalytique responsable de la dégradation. Elles sont appelées des CAZYmes. En ce qui concerne les autres constituants de la paroi végétale (lignines, extractibles, cires,…), certaines Glutathion Transférases peuvent réagir avec certains de ces composés. L’idée globale de PROTCAR était donc d’utiliser ces enzymes (ou des modules les constituant) comme outils de caractérisation de la paroi végétale
Objectifs et Démarche — L’objectif principal de ce projet consistait à valider la démarche proposée en produisant différents modules protéiques couplés à un module fluorescent. Différentes enzymes/modules ont choisis en fonction de leur activité/affinité envers différents constituants de la paroi végétale. Ces modules ont été testés afin de valider l’hypothèse de départ en utilisant des techniques de microscopie et de spectroscopie à fluorescence.
Résultats marquants —
- Les protéines d’origine bactérienne LigE, LigF, CBM3a et la protéine fongique PcGSTFuA ont été produites et purifiées avec ou sans fusion mcherry.
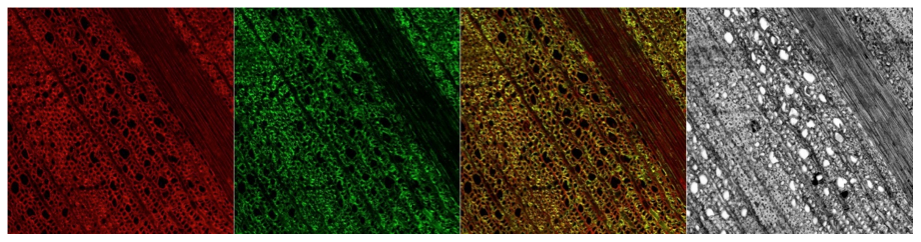
- En collaboration avec la plateau xyloscience et en utilisant les outils de microscopie de la plateforme écogénomique, une méthode permettant de visualiser et de quantifier l’adhésion des protéines testées à des coupes de chêne a été développée
- Sur coupes de chêne, après numérisation des observations par microscopie confocale, on mesure quantitativement et significativement une adhésion différente des différentes protéines testées. Ces résultats sont en accord avec la composition du bois
- En utilisant ces modules protéiques, une modification chimique de fibres de lin, de chanvre et de bambou a pu être mise en évidence (amélioration de l’hydrophobicité)
Conclusion et Perspectives —
Ce programme visait à mettre au point des outils enzymatiques permettant à la fois la caractérisation et la modification de parois lignocellulosiques et plus particulièrement. Ces outils sont actuellement utilisés au sein de différents programmes de recherche. Cette méthodologie sera notamment utilisée dans le cadre du programme NEWFIBRE développé avec les partenaires ayant collaboré sur ce programme : la société BIOLIE, le CETELOR et le LERMAB. Le programme NEWFIBRE financé par le FEDER et la Région Lorraine vise à développer une nouvelle filière « fibre » régionale et regroupe une grande partie des industries textiles présentes en Lorraine. Une valorisation de ces travaux sous forme d’un brevet est à l’étude.